이번 글에서는 Semi-supervised classification with Graph Convolutional Networks - Thomas N. Kipf, Max Welling 라는 논문에서 제시하는 GCN model의 코드를 분석해보고자 한다. 이론적 내용은 논문을 참고하자.
이 논문의 목적은 거대한 그래프에서 노드들 중 아주 일부만 레이블이 달려있을 때 노드를 잘 분류하는 모델을 만드는 것이다. - graph based semi supervised learning
간단하게(?) GCN을 설명하자면 graph에서 node들 중 edge로 연결된 node들 끼리만 연산을 하는 Neural Network이다. 이는 아래의 식으로 쉽게 표현가능하다.

여기서 A는 인접행렬을 의미하고, 가장 아래의 식에서 D는 각 노드의 차수가 diagonal element인 대각행렬을 의미한다.
일반적으로 image에 대한 학습을 할 때 전체 image가 아닌 image의 여러 부분들을 나누어 추상화한다.(하나의 사진에 꽃, 구름, 해가 모두 담겨 있을 수도 있다.) 따라서, FC를 쓰지 않고 Convolutional Neural Network를 이용한다.
GCN은 Convolution이라는 단어가 들어간 만큼 edge로 연결된 node들 간의 연산을 집중적으로 수행하게된다.
내가 GCN에 관심을 가지게 된 이유는 소셜 네트워크 분석을 해보고 싶기 때문이다. node를 다양한 node feature를 지닌 사람으로 보고 edge를 소셜네트워크에서의 연결성으로 본다면, 우리는 GCN을 통해 집단적 feature를 뽑아낼 수 있으며, 이를 이용해 다양한 classification을 진행할 수 있다.
설명은 간단히하고 Pytorch 프레임워크를 이용한 GCN 실습코드를 보자.

|
1
2
3
4
5
6
7
8
9
10
|
'''
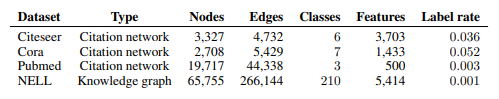
데이터 설명 in English ind.dataset_str.x = the feature vectors of the training instances as scipy.sparse.csr.csr_matrix object; ind.dataset_str.tx = the feature vectors of the test instances as scipy.sparse.csr.csr_matrix object;
ind.dataset_str.allx = the feature vectors of both labeled and unlabeled training instances (a superset of ind.dataset_str.x) as scipy.sparse.csr.csr_matrix object;
ind.dataset_str.y = the one-hot labels of the labeled training instances as numpy.ndarray object;
ind.dataset_str.ty = the one-hot labels of the test instances as numpy.ndarray object;
ind.dataset_str.ally = the labels for instances in ind.dataset_str.allx as numpy.ndarray object;
ind.dataset_str.graph = a dict in the format {index: [index_of_neighbor_nodes]} as collections.defaultdict object;
ind.dataset_str.test.index = the indices of test instances in graph, for the inductive setting as list object.
''' |
cs |
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
126
127
128
129
130
131
132
133
134
135
136
137
138
139
140
|
"""
gcn/data directory에서 가져올 데이터 설명
ind.dataset_str.x => scipy.sparse.csr.csr_matrix object로 된 트레이닝을 위한 feature vectors
ind.dataset_str.tx => scipy.sparse.csr.csr_matrix object로 된 테스트를 위한 feature vectors
ind.dataset_str.allx => scipy.sparse.csr.csr_matrix object로 된, 트레이닝에 사용할 레이블이 있는 feature vectors와 레이블이 없는 feature vectors(ind.dataset_str.x를 포함하는 집합)
ind.dataset_str.y => 레이블이 있는 트레이닝 데이터에 대한 레이블(numpy.ndarray object, 원핫인코딩)
ind.dataset_str.ty => 레이블이 있는 테스트 데이터에 대한 레이블(numpy.ndarray object, 원핫인코딩)
ind.dataset_str.ally => ind.dataset_str.allx의 레이블을 담고있다. (numpy.ndarray object)
ind.dataset_str.graph => {index: [index_of_neighbor_nodes]} 형태로 graph representation을 하는 collections.defaultdict object
ind.dataset_str.test.index => 그래프에서 테스트 데이터의 인덱스들, for the inductive setting as list object
* 위의 데이터들은 모두 pickle module을 이용해 파싱해야한다 *
* 디렉토리에 있는 여러 그래프 데이터 중 여기서 이용할 데이터는 pubmed dataset이다. *
"""
import numpy as np
import pickle as pkl #serialize를 위한 바이너리 프로토콜 구현. 클래스를 통째로 저장 및 불러올 때 유용하다.
import networkx as nx #그래프를 다루기 위한 파이썬 패키지
import scipy.sparse as sp #행렬의 대부분의 원소가 0인 sparse matrix를 만들기 위해 불러온다
from scipy.sparse.linalg.eigen.arpack import eigsh #대칭행렬에서 k개의 eigen vector와 eigen value를 가져옴
import sys
import torch
from torch import nn
from torch import optim
from torch.nn import functional as F
import numpy as np
import easydict
#easydict를 이용해 hyperparameter 및 사용할 데이터를 미리 정의한다.
args = easydict.EasyDict({'dataset': 'pubmed',
'model': 'gcn',
'learning_rate': 0.01,
'epochs': 400,
'hidden': 16,
'dropout': 0.5,
'weight_decay' : 5e-4,
'early_stopping':10,
'max_degree' : 3
})
names = ['x', 'y', 'tx', 'ty', 'allx', 'ally', 'graph']
objects = []
for i in range(len(names)): #각 파일들을 열고 pickle모듈을 이용해 serialized된 데이터를 파싱한다.
with open("data/ind.{}.{}".format(args.dataset, names[i]), 'rb') as f:
objects.append(pkl.load(f, encoding='latin1'))
print(len(objects)) #7이 잘 나온다.
def parse_index_file(filename): #테스트데이터의 index를 담고 있는 파일을 파싱하기 위한 함수
index = []
for line in open(filename):
index.append(int(line.strip()))
return index
x, y, tx, ty, allx, ally, graph = tuple(objects) #튜플로 변환하고 7개의 데이터를 나눈다.
test_idx_reorder = parse_index_file("data/ind.{}.test.index".format(args.dataset))
test_idx_range = np.sort(test_idx_reorder) #테스트데이터의 index를 오름차순 정렬한다.
features = sp.vstack((allx, tx)).tolil() #train데이터와 테스트데이터를 하나의 matrix로 결합하고 LInked List format으로 변환
features[test_idx_reorder, :] = features[test_idx_range, :] #테스트 데이터의 인덱스를 이용해 테스트 데이터를 전체 메트릭스 안에서 재배치 한다
adj = nx.adjacency_matrix(nx.from_dict_of_lists(graph)) #node list를 받아 adjacency matrix를 만들어주는 함수이다.
labels = np.vstack((ally, ty)) # 전체 레이블 데이터를 하나의 matrix로 만든다.
labels[test_idx_reorder, :] = labels[test_idx_range, :] #테스트데이터의 인덱스를 이용해 테스트 레이블 데이터를 전체 레이블 matrix에서 재배치한다
idx_test = test_idx_range.tolist()
idx_train = range(len(y))
idx_val = range(len(y), len(y)+500)
'''
이렇게 train, test, validation 데이터 나누기를 위한 준비가 완성되었다.
'''
def sample_mask(idx, l): #마스크를 만드는 함수
#주어진 데이터가 마스킹이 되어있지 않은 상태이기 때문에 따로 마스킹이 필요하다. mask = np.zeros(l) # ㅣ차원의 벡터를 만들고 0으로 채운다.
mask[idx] = 1 # 0벡터의 idx 자리에 1을 넣는다. 0은 마스킹을 하는 것이고 1은 마스킹을 하지 않는 것이다. return np.array(mask, dtype=np.bool)
num_of_labels = labels.shape[0] #19717개의 레이블이 있다.
train_mask = sample_mask(idx_train, num_of_labels)
val_mask = sample_mask(idx_val, num_of_labels)
test_mask = sample_mask(idx_test, num_of_labels)
y_train = np.zeros(labels.shape) #labels.shape는 (19717, 3) 이다.
y_val = np.zeros(labels.shape)
y_test = np.zeros(labels.shape)
#label data를 마스킹한 후 재구성한다. Semi-Supervised이기 때문에 마스킹을 해주는 것이다.
y_train[train_mask, :] = labels[train_mask, :]
y_val[val_mask, :] = labels[val_mask, :]
y_test[test_mask, :] = labels[test_mask, :]
seed = 123
np.random.seed(seed)
torch.random.manual_seed(seed)
#이제 차원이 제대로 맞추어져 있는지 확인해보자
print('adj:', adj.shape)
print('features:', features.shape)
print('y:', y_train.shape, y_val.shape, y_test.shape)
print('mask:', train_mask.shape, val_mask.shape, test_mask.shape)
|
cs |
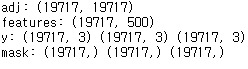
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
|
#GCN 모델에 데이터를 넣기 위해서는 데이터를 알맞은 형태로 만들어주는 전처리가 필요하다. 아래는 이를 위한 함수들이다.
def to_tuple(mx): #matrix를 좌표, 값, shape 형태의 튜플로 변환해주는 함수
if not sp.isspmatrix_coo(mx): # mx가 coo_matrix type인지 True/False로 반환해준다
mx = mx.tocoo() # coordinate(좌표) 형태의 sparse matrix로 변환한다.
# https://matteding.github.io/2019/04/25/sparse-matrices/ 에서 coo_matrix 형태의 행렬을 확인할 수 있다.
coords = np.vstack((mx.row, mx.col)).transpose() #좌표
values = mx.data #좌표에 할당되는 값
shape = mx.shape
return coords, values, shape
def sparse_to_tuple(sparse_mx): #sparse matrix 를 tuple representation으로 변환한다.
if isinstance(sparse_mx, list):
for i in range(len(sparse_mx)):
sparse_mx[i] = to_tuple(sparse_mx[i])
else:
sparse_mx = to_tuple(sparse_mx)
return sparse_mx
def preprocess_features(features):
"""
feature matrix를 행 별로 정규화하고 tuple representation으로 변환한다.
"""
rowsum = np.array(features.sum(1)) # 각 행의 합을 구한다. [2708, 1]
r_inv = np.power(rowsum, -1).flatten() # np.power()의 두번째 arg에 -1을 줘서 1/rowsum으로 만들고 flatten한다, [2708]
r_inv[np.isinf(r_inv)] = 0. # infinite data를 0으로 바꿔준다.
r_mat_inv = sp.diags(r_inv) # sparse diagonal matrix를 만든다, [2708, 2708]
features = r_mat_inv.dot(features) # D^-1 를 구한다:[2708, 2708]@X:[2708, 2708]
return sparse_to_tuple(features) # [coordinates, data, shape], []
def normalize_adj(adj):
"""
adjacency matrix를 Symmetrically normalize
노드의 연결성을 차원(degree)라고 부르는데, 인접 행렬 A의 차원(D)에 대한 역행렬을 곱해주면 정규화를 할 수 있다.
"""
adj = sp.coo_matrix(adj)
rowsum = np.array(adj.sum(1)) # D
d_inv_sqrt = np.power(rowsum, -0.5).flatten() # D^-0.5
d_inv_sqrt[np.isinf(d_inv_sqrt)] = 0.
d_mat_inv_sqrt = sp.diags(d_inv_sqrt) # D^-0.5
return adj.dot(d_mat_inv_sqrt).transpose().dot(d_mat_inv_sqrt).tocoo() # D^-0.5 A D^0.5 를 만들어 준다.
def preprocess_adj(adj):
"""
위에 만든 normalize_adj를 이용해서 A + I 행렬을 정규화하고 tuple representation으로 변환하는 함수를 만든다.
"""
adj_normalized = normalize_adj(adj + sp.eye(adj.shape[0]))
return sparse_to_tuple(adj_normalized)
|
cs |
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
126
127
128
129
130
131
132
133
134
135
136
137
138
139
140
141
142
143
144
145
146
147
148
149
150
151
152
153
154
155
156
157
158
159
160
161
162
|
def masked_loss(out, label, mask):
'''
loss function으로 이용되는 함수이다. mask 벡터의 원소가 True면 1 False면 0이기 때문에 True인 부분의 loss만을 가중치를 주어 계산하고,
0인 부분, 즉 False인 부분은 masking이 된 것으로 판단하여 loss 계산에서 제외한다.
'''
loss = F.cross_entropy(out, label, reduction='none') #우선 레이블이 잇는 데이터의 아웃풋과 레이블의 크로스엔트로피로스를 구한다.
mask = mask.float() #0,1 (False, True의 int형)로 이루어진 벡터로 되어있는 mask를 float으로 바꿔준다.
mask = mask / mask.mean() #벡터의 각 값을 mask의 평균으로 나눈다.
loss *= mask #mask 벡터에 loss를 곱한다.
loss = loss.mean()
return loss
def masked_acc(out, label, mask):
'''
accuracy를 구하는 함수이다. 위와 마찬가지로 마스킹이 되지 않은 부분, 즉 마스크 벡터가 True인 부분의 accuracy를 구한다.
'''
# [node, f]
pred = out.argmax(dim=1)
correct = torch.eq(pred, label).float()
mask = mask.float()
mask = mask / mask.mean()
correct *= mask
acc = correct.mean()
return acc
def sparse_dropout(x, rate, noise_shape):
'''
drop-out을 시행하는 함수이다. input matrix와 dropout의 비율과 0이 아닌 값을 가지는 원소의 개수를 argument로 받는다.
'''
random_tensor = 1 - rate
random_tensor += torch.rand(noise_shape).to(x.device)
dropout_mask = torch.floor(random_tensor).byte()
i = x._indices() # [2, 49216]
v = x._values() # [49216]
# [2, 49216] => [49216, 2] => [remained node, 2] => [2, remained node]
i = i[:, dropout_mask]
v = v[dropout_mask]
out = torch.sparse.FloatTensor(i, v, x.shape).to(x.device)
out = out * (1./ (1-rate))
return out
class GraphConvolution(nn.Module): #graph convolution을 실행할 함수를 만든다.
def __init__(self, input_dim, output_dim, num_features_nonzero,
dropout=0.,
is_sparse_inputs=False,
bias=False,
activation = F.relu,
featureless=False):
super(GraphConvolution, self).__init__()
self.dropout = dropout
self.bias = bias
self.activation = activation
self.is_sparse_inputs = is_sparse_inputs
self.featureless = featureless
self.num_features_nonzero = num_features_nonzero
self.weight = nn.Parameter(torch.randn(input_dim, output_dim)) # weight parameter matrix를 차원에 맞춰 랜덤한 수를 넣어 만든다.
self.bias = None
if bias:
self.bias = nn.Parameter(torch.zeros(output_dim)) # bias parameter matrix를 차원에 맞춰 0을 패딩하여 만든다.
def forward(self, inputs):
'''
forward()는 모델이 학습데이터를 입력받아서 forward propagation을 진행시키는 함수이고, 반드시 forward 라는 이름의 함수이어야 한다.
forward()는 __call__의 역할을 수행하기 때문에, 인스턴스를 만들면 바로 실행된다. model.forward(inputs)가 아닌 model(inputs)로 forward를 진행한다.
'''
x, support = inputs # support는 classification을 할 때 중심이 되는 벡터
if self.training and self.is_sparse_inputs:
x = sparse_dropout(x, self.dropout, self.num_features_nonzero)
elif self.training:
x = F.dropout(x, self.dropout)
# convolve
if not self.featureless: # if it has features x
if self.is_sparse_inputs:
xw = torch.sparse.mm(x, self.weight) # sparse matrix 형태의 input * weight
else:
xw = torch.mm(x, self.weight)
else:
xw = self.weight
out = torch.sparse.mm(support, xw)
if self.bias is not None:
out += self.bias # input * weight + bias
return self.activation(out), support
class GCN(nn.Module):
def __init__(self, input_dim, output_dim, num_features_nonzero):
super(GCN, self).__init__()
self.input_dim = input_dim # 1433
self.output_dim = output_dim
print('input dim:', input_dim)
print('output dim:', output_dim)
print('num_features_nonzero:', num_features_nonzero)
self.layers = nn.Sequential(GraphConvolution(self.input_dim, args.hidden, num_features_nonzero,
activation=F.relu,
dropout=args.dropout,
is_sparse_inputs=True),
GraphConvolution(args.hidden, output_dim, num_features_nonzero,
activation=F.relu,
dropout=args.dropout,
is_sparse_inputs=False),
)
def forward(self, inputs):
x, support = inputs
x = self.layers((x, support))
'''
inputs라는 하나의 arg를 받기때문에 x, support를 튜플로 묶어서 input으로 넣고 GraphConvoltion class 의 forward를 실행시킨다.
'''
return x
def l2_loss(self): #weight^2을 regularization으로 준다.
layer = self.layers.children() # gives an iterator over the layers in network model.
layer = next(iter(layer)) #레이어를 iterate하게 꺼낸다.
loss = None
for p in layer.parameters(): # 레이어의 weight를 제곱하여 loss에 더한다.
if loss is None:
loss = p.pow(2).sum()
else:
loss += p.pow(2).sum()
return loss
|
cs |
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
|
#모델에 넣기 위한 데이터를 준비해보자
features = preprocess_features(features) # [49216, 2], [49216], [2708, 1433]
supports = preprocess_adj(adj) #normalize하고 tuple with coordination 형태로 나타낸 adjacency matrix
device = torch.device('cuda') #cuda 이용을 위해 미리 선언
train_label = torch.from_numpy(y_train).long().to(device)
num_classes = train_label.shape[1]
train_label = train_label.argmax(dim=1)
train_mask = torch.from_numpy(train_mask.astype(np.int)).to(device)
val_label = torch.from_numpy(y_val).long().to(device)
val_label = val_label.argmax(dim=1)
val_mask = torch.from_numpy(val_mask.astype(np.int)).to(device)
test_label = torch.from_numpy(y_test).long().to(device)
test_label = test_label.argmax(dim=1)
test_mask = torch.from_numpy(test_mask.astype(np.int)).to(device) #False가 18717개, True가 1000개
i = torch.from_numpy(features[0]).long().to(device)
v = torch.from_numpy(features[1]).to(device)
feature = torch.sparse.FloatTensor(i.t(), v, features[2]).to(device)
i = torch.from_numpy(supports[0]).long().to(device)
v = torch.from_numpy(supports[1]).to(device)
support = torch.sparse.FloatTensor(i.t(), v, supports[2]).float().to(device)
print('x :', feature)
print('sp:', support)
num_features_nonzero = feature._nnz() #nnz는 행렬에 포함된 0이 아닌 요소 개수이다. number of non-zeros
feat_dim = feature.shape[1]
net = GCN(feat_dim, num_classes, num_features_nonzero)
net.to(device)
optimizer = optim.Adam(net.parameters(), lr=args.learning_rate) #Adam Optimizer 이용
#이제 학습을 시작해보자
net.train()
for epoch in range(args.epochs):
out = net((feature, support))
out = out[0]
loss = masked_loss(out, train_label, train_mask)
loss += args.weight_decay * net.l2_loss() # weight decay * l2_loss를 loss에 추가한다.
acc = masked_acc(out, train_label, train_mask)
optimizer.zero_grad()
loss.backward()
optimizer.step()
if epoch % 10 == 0:
print(epoch, loss.item(), acc.item())
net.eval()
out = net((feature, support))
out = out[0]
acc = masked_acc(out, test_label, test_mask)
print('test:', acc.item())
|
cs |

코드참고 : github.com/dragen1860/GCN-PyTorch
dragen1860/GCN-PyTorch
Graph Convolution Network for PyTorch. Contribute to dragen1860/GCN-PyTorch development by creating an account on GitHub.
github.com
'deeplearning' 카테고리의 다른 글
| CRF를 이용한 Named Entity Recognition (0) | 2020.09.18 |
|---|---|
| Explainable AI - Integrated Gradients (IG) (0) | 2020.08.26 |
| Optimization - Momentum, RMSProp, Adam (0) | 2020.08.21 |
| Gensim Word2Vec Fine-tuning (1) | 2020.06.22 |
| (연습)Multilingual-BERT를 이용해 크롤링한 영화 댓글 감성 분류 하기 #COLAB (0) | 2020.04.20 |



